Em uma realidade onde diversas campanhas contra o câncer infantil no Brasil transmitem mensagens de esperança para um futuro com mais cura, o meduloblastoma entra em evidência. Classificado como um tumor cerebral agressivo que acomete crianças, a limitação dos métodos atuais para prever a evolução da doença resulta em um terço dos pacientes não alcançando a sobrevivência a longo prazo. No entanto, uma recente descoberta científica abre novas possibilidades: dois genes identificados no tumor podem servir como marcadores biológicos promissores para classificar pacientes em grupos de risco e indicar tratamentos mais adequados, oferecendo uma expectativa clínica mais otimista.
A pesquisa, premiada em 1º lugar na categoria Médicos durante o 1º Congresso Internacional de Oncologia Pediátrica do Grupo de Apoio ao Adolescente e à Criança com Câncer (GRAACC), ressalta a necessidade de métodos mais eficazes para a estratificação de risco. Os pesquisadores identificaram os genes-chave Cell Migration-Inducing Protein(CEMIP) e Nuclear Cap Binding Protein 3(NCBP3) como biomarcadores promissores. Segundo a coordenadora do projeto, Marialva Sinigaglia, do Instituto do Câncer Infantil (ICI), no Rio Grande do Sul, pacientes com níveis elevados desses genes no meduloblastoma sobrevivem por mais tempo comparado aos pacientes com menor nível.

Para encontrar o grupo de genes que pudesse prever o comportamento do câncer, Marialva relata que foram utilizados dados de mais de 1.600 pacientes com o tumor, disponíveis em bancos de dados públicos. Os métodos e testes estatísticos empregados para averiguar o valor prognóstico da assinatura genética proposta confirmaram a alta capacidade preditiva do modelo, conforme relato da doutora.
A pesquisa utilizou a técnica Identificação de Genes Dirigidos por Metilação (MDGs) para identificar os genes, que juntos formaram a assinatura capaz de prever a evolução do tumor. “Para isso, utilizamos um modelo estatístico de aprendizado de máquina, a regressão de LASSO”, conta o pesquisador Gustavo Lovatto Michaelsen, do Programa de Pós-graduação em Bioinformática (PPg-Bioinfo) da UFRN. O modelo demonstrou a capacidade de identificar pacientes em grupos de diferentes níveis de risco, oferecendo uma ferramenta prognóstica mais confiável em comparação com parâmetros clínicos tradicionais.

De acordo com Gustavo, a capacidade preditiva foi avaliada através de análises estatísticas robustas, como a regressão de Cox, técnica utilizada para analisar dados de sobrevivência, e análise de curvas de Receiver Operating Characteristic (ROC), técnica que avalia a capacidade de um modelo preditivo em classificar corretamente os eventos de interesse, como sobrevivência ou morte, em diferentes princípios de probabilidade.
O pesquisador reforça que, ao analisar a atividade de um grupo de genes em amostras do tumor, será possível classificá-los em diferentes grupos de risco para prever a evolução da doença e direcionar pacientes para tratamentos mais adequados com base em sua biologia. Além disso, a pesquisa aponta que a abordagem por biomarcadores, como a GS-2, tem impacto significativo na sobrevida global e na minimização de efeitos colaterais.
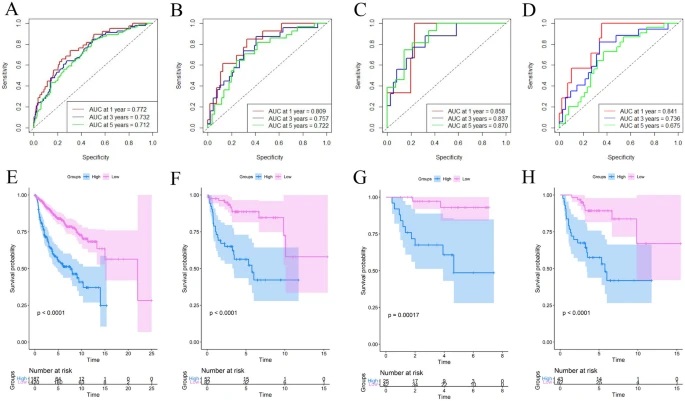
Mesmo com resultados promissores, Marialva ressalta que a pesquisa é um trabalho inicial, sem validação em bancada ou pacientes. “É necessário realizar estudos clínicos prospectivos e multicêntricos para confirmar a utilidade clínica desse modelo e avaliar seu impacto no tratamento de pacientes com esta doença”, conclui.
O trabalho, publicado no periódico Journal of Molecular Neuroscience, é uma parceria entre a UFRN e o ICI, premiado com o título de melhor ONG do Brasil em 2023 pelo Prêmio Melhores ONGs. Além disso, o PPg-Bioinfo teve apoio do Núcleo de Processamento de Alto Desempenho (NPAD/UFRN) no processamento e análise de dados tumorais.
Fonte: Agecom/UFRN












































































