Apenas no Brasil, o SARS-CoV-2 desencadeou 660 mil mortes e cerca de 29,9 milhões infecções. Com o propósito de descobrir novas alternativas de tratamento para a covid-19 grave, novo estudo, publicado em preprint, identificou mais de 14 novas alterações genéticas ligadas ao estágio crítico da doença, capazes de contribuir para o desenvolvimento de novos medicamentos e alvos terapêuticos para pacientes hospitalizados ou que morreram em virtude da patologia. A pesquisa integra o trabalho Genetics Of Mortality in Critical Care (GenOMICC) e analisa dados dos consórcios Coronavirus Clinical Characterisation Consortium (ISARIC4C) e Spanish Coalition to Unlock Research on host genetics on covid-19 (SCOURGE), sendo este último associado à UFRN.
André Ducati Luchessi, professor do Departamento de Análises Clínicas e Toxicológicas (DACT/UFRN), um dos autores do estudo, observa que o uso de resultados de múltiplos consórcios internacionais é algo inédito na literatura científica. “A ligação entre esses consórcios permite que o grupo do professor Kenneth Baillie, da Universidade de Edimburgo, junto com outros pesquisadores, como Dr. Esteban Parra e sua equipe da Universidade de Toronto, possam utilizar essas informações de forma conjunta e encontrar novos resultados. É uma abordagem de análises de dados em larga escala baseado em Machine Learning, (tecnologia na qual os computadores aprendem conforme a associação de dados)”, afirma.
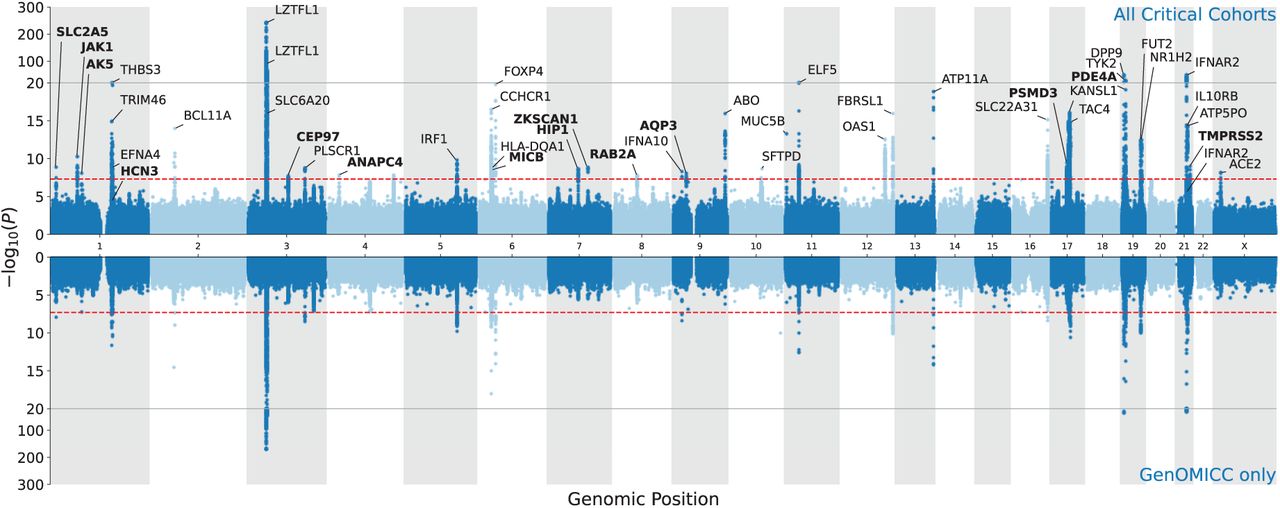
O GenÔMICO combinou dados de genótipos de microarranjos de 11.325 casos graves, sendo 9.279 casos do Reino Unido e 2.186 do Brasil. Em relação ao ISARC e o SCOURGE, foram associados dados de 6.589 casos de pacientes hospitalizados em estágio de risco. Para investigação das informações, o trabalho utilizou a metanálise de dados – metodologia estatística que reúne resultados de dois ou mais estudos sobre um mesmo objeto de pesquisa – e a técnica do Estudos de Associação Ampla do Genoma (GWAS, na sigla em inglês), que consiste em identificar associações entre características genômicas e traços fenotípicos para o estudo de doenças.
A escolha dos grandes consórcios foi uma estratégia fundamental para a metanálise, pois ela exige intersecções entre os dados que serão avaliados. Essa característica, conforme aponta o pesquisador, é algo encontrado nas organizações escolhidas, uma vez que todos se debruçam na ligação de dados clínicos de hospitalização ou gravidade com as alterações encontradas no DNA dos pacientes infectados pelo Sars-Cov-2. No caso do trabalho da UFRN junto ao Scourge, sob a coordenação da professora Vivian Nogueira Silbiger (DACT/UFRN), afirma que foi fundamental para a adição de amostras de pacientes com resultados positivos da Sars-Cov-2 no grupo.
Contribuições científicas e limitações
A conexão entre alterações genéticas com os genes causais da doença é um dos diferenciais do estudo. Isso acontece porque muitas pesquisas analisam apenas as mudanças nos genes de quadros positivos para covid-19, mas é mediante as associações genéticas que as chances de encontrar genes associados ao fenótipo do estágio grave da patologia são elevados. No estudo, André Luchessi observa que os resultados sinalizam para um grupo de genes ligados ao processo inflamatório, algo clinicamente conhecido, mas agora é possível nomear os genes encontrados e, na sequência, propor novas possibilidades de tratamento para a doença.
Embora o estudo aumente as chances de descoberta para novas medidas de tratamento, ele combina sinais genéticos para vários estágios na progressão da doença, como exposição viral, infecção, replicação e desenvolvimento de doença pulmonar inflamatória. Em outras palavras, não é possível identificar em qual fase do avanço da patologia ou em que região do corpo do paciente as manifestações dos genes causais acontecem. Outra limitação é a ausência de diversidade dos indivíduos analisados, visto que eles são, majoritariamente, de descendência europeia, apontando a necessidade de ampliar a participação de outros grupos em próximas pesquisas.
Luchessi ressalta que a falta de dados efetivamente experimentais e capazes de associar os genes sinalizados com a gravidade da covid-19 compromete a maior precisão da pesquisa. “Resumidamente, utilizamos uma estratégia de genotipagem em larga escala para determinar os genótipos dos pacientes e, posteriormente, meta-analizados com outros dados já disponíveis, como é o caso do SCOURGE e ISARIC4C, finalizando com a chamada TWAS (Análise de transcriptômica completa). A nossa expectativa é, primeiro, publicar esses achados em uma revista de alto impacto e seguir para uma validação experimental das participações destes genes (encontrados na pesquisa) com a doença”, conclui.
Fonte: Agecom/UFRN


